Présentation
Pour plus d’infos sur notre équipe et notre recherche, visitez notre site web: https://www.legubelab.com/

Visitez la page web de Raphaël Mourad ici.
Projets
Notre ADN subit de nombreuses agressions externes qui mènent à des dommages menaçant l’intégrité du génome. Parmi ces dommages, les cassures double-brin de l’ADN (DSB) sont les plus délétères puisqu'elles peuvent conduire à diverses mutations et réarrangements chromosomiques, événements à l’origine de cancers. Les DSB surviennent au cours du développement dans le cadre de processus programmés (lors de la formation du système immunitaire, par exemple), mais également en raison de divers stress environnementaux (fumée de cigarette, irradiation, etc). Au cours des dernières années, il est devenu évident que la chromatine, qui encapsule l'ADN dans les noyaux et qui contient les informations épigénétiques, joue un rôle déterminant dans la réparation de l'ADN. La réparation des DSB dans le contexte chromatinien soulève donc plusieurs questions auxquelles nous essayons de répondre au laboratoire.
Pour répondre à ces questions, nous avons développé la lignée cellulaire DIvA, qui est un système cellulaire expérimental puissant qui permet la création de DSB à des positions bien connues du génome et dans divers contextes chromatiniens. Cette lignée cellulaire permet d’induire de nombreuses DSB, et présente donc l’avantage d’avoir un pouvoir statistique sur une large gamme de DSB.
Plus précisément, ce modèle cellulaire se base sur l'expression d'une enzyme de restriction (AsiSI) fusionnée à un domaine de liaison du récepteur aux oestrogènes qui répond à la drogue hydroxy-tamoxifène (OHT). Le traitement OHT de cette lignée cellulaire déclenche la localisation nucléaire de l'enzyme de restriction AsiSI et donc l'induction rapide (<1 heure) de multiples DSB spécifiques à une séquence, répandues dans le génome (Iacovoni et al, 2010; Massip et al, 2010). Nous possédons également une deuxième lignée cellulaire, appelée AID-DIvA, dont AsiSI est couplée au système AID (auxin-inducible degron) permettant sa dégradation rapide après l’ajout d’auxine, ce qui permet la réparation des DSB (Aymard et al, 2014). En d’autres termes, ce système permet une induction des DSB sur des sites annotés, dispersés sur le génome humain.

Comment l'état de la chromatine influence-t-il la reconnaissance des DSB et le choix de la voie de réparation ?
Chez les eucaryotes, la réparation des DSB se produit au sein de la chromatine. Un bon nombre d'études, y compris la nôtre, suggère que la chromatine joue un rôle important dans le choix de la voie de la réparation. Notre hypothèse de travail est qu'il existe un "code histone de la réparation". En d’autres mots, la chromatine influencerait la réparation des DSB à différentes étapes, de la détection de la DSB à la récupération de séquence. En effet il est possible que les marques d'histones présentes avant l'apparition des DSB aide spécifiquement à recruter et/ou à stabiliser l'une ou l'autre voie de réparation (Clouaire, et al, 2015). Nous avons en effet montré que la marque histone H3K36me3 est importante pour canaliser la voie de réparation des DSB induites dans les gènes actifs vers la HR (Aymard et al, 2014). De plus, la chromatine peut également être modifiée pendant le processus de réparation, et ce à différentes échelles, d'une manière qui est liée à la voie de réparation utilisée (Clouaire et al. 2018).
Afin de déchiffrer le paysage chromatinien induit par les DSB, nous utilisons principalement les techniques de BLESS, ChIP-chip / seq et ARN seq. Ces techniques nous permettent d’obtenir les profils des modifications de la chromatine et des complexes de réparation autour des DSB et ce à haute résolution (Iacovoni et al, 2010; Massip et al, 2010; Caron et al, 2012; Clouaire et al, 2018).
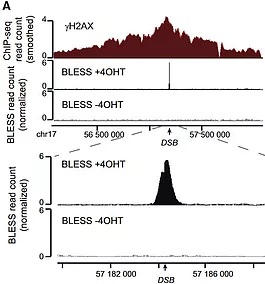
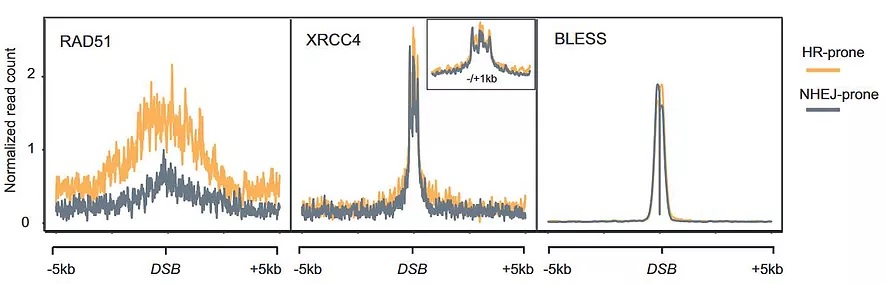
Comment les gènes actifs sont-ils réparés ?
Des travaux récents ont révélé que de manière surprenante les loci transcriptionnellement actifs sont fréquemment sujets à des DSB. Nos travaux suggèrent que les gènes actifs endommagés manifestent un comportement très particulier par rapport au reste du génome : ils subissent un retard de réparation important et se regroupent en « cluster » via un mécanisme qui dépend de l’actine/LINC et du complexe MRN en G1 (Aymard et al, 2017), alors qu'ils sont principalement réparés par recombinaison homologue d'une manière dépendante de la sénataxine dans des cellules post-réplicatives (Cohen et al, 2018). Notre équipe essaie de caractériser plus en détail cette nouvelle voie de réparation des DSB que nous appelons « transcription-coupled DSB repair » (TC-DSBR) (Marnef et al, 2018).
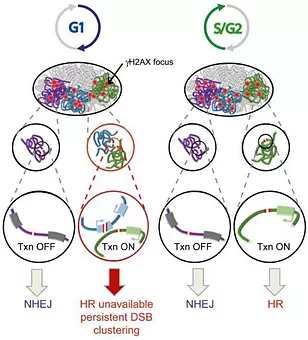
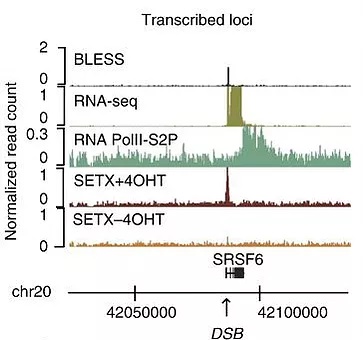
Comment la conformation chromosomique change-t-elle après cassures ?
Nos travaux ont permis de caractériser le paysage chromatinien (modifications d'histones) induites par les DSB. Cependant, la structure de la chromatine (densité de nucléosomes, repliement) en cis autour du DSB reste à déterminer. À l'aide des techniques de capture de la conformation chromosomique, nous essayons de comprendre la structure en 3D des chromosomes après DSB (Aymard et al. 2017). En effet, le Capture-HiC nous a permis de comprendre à l'échelle du génome les contacts longue distance de l’ADN, et a notamment dévoilé le regroupement en « cluster » des DSB se produisant au niveau des gènes actifs.

EQUIPE LEGUBE 2023

Publications
- Mazzocca M, Loffreda A, Colombo E, Fillot T, Gnani D, Falletta P, Monteleone E, Capozi S, Bertrand E, Legube G, Lavagnino Z, Tacchetti C, Mazza D.
Chromatin organization drives the search mechanism of nuclear factors
Nature Communications
2023 Oct - Arnould C, Rocher V, Saur F, Bader AS, Muzzopappa F, Collins S, Lesage E, Le Bozec B, Puget N, Clouaire T, Mangeat T, Mourad R, Ahituv N, Noordermeer D, Erdel F, Bushell M, Marnef A, Legube G..
Chromatin compartmentalization regulates the response to DNA damage
Nature
2023 Oct - Gritti I, Basso V, Rinchai D, Corigliano F, Pivetti S, Gaviraghi M, Rosano D, Mazza D, Barozzi S, Roncador M, Parmigiani G, Legube G, Parazzoli D, Cittaro D, Bedognetti D, Mondino A, Segalla S, Tonon G.
Loss of ribonuclease DIS3 hampers genome integrity in myeloma by disrupting DNA:RNA hybrid metabolism.
EMBO Journal
2022 Nov - Groelly FJ, Dagg RA, Petropoulos M, Rossetti GG, Prasad B, Panagopoulos A, Paulsen T, Karamichali A, Jones SE, Ochs F, Dionellis VS, Puig Lombardi E, Miossec MJ, Lockstone H, Legube G, Blackford AN, Altmeyer M, Halazonetis TD, Tarsounas M.
Mitotic DNA synthesis is caused by transcription-replication conflicts in BRCA2-deficient cells
Mol Cell
2022 Sep - Cohen S, Guenolé A, Lazar I, Marnef A, Clouaire T, Vernekar DV, Puget N, Rocher V, Arnould C, Aguirrebengoa M, Genais M, Firmin N, Shamanna RA, Mourad R, Bohr VA, Borde V, Legube G..
A POLD3/BLM dependent pathway handles DSBs in transcribed chromatin upon excessive RNA:DNA hybrid accumulation.
Nature Communication
2022 Apr
Voir toutes les publications
Financements













Anciens membres

































